はじめに
少しご無沙汰してしまい、また読者様から重くないやつを定期的にと助言いただきましたので、少し短めの投稿を記事作成のリハビリもかねてしてみようと思います
今回はそもそもこのブログで扱っているモデリングとは何だったのか?について改めて書いてみようと思います。 さらに今後の記事では続きとしてモデル選択の基準、特にベイズで利用できるWAIC(広く使える情報量基準)について書いてみようと思っています。 ただし、難しいことまではよくわからないので、感覚で理解できているところまで。
モデリング
モデリングとは
そもそもの話です。
統計解析ではデータが生成されるメカニズムを組み立てることでデータの解釈を進めていきます。このような活動をモデリングと呼んでいます。ただし、あるデータに対する数理モデルの候補は通常一つではありません。
例として、今回は回帰分析等のサンプルデータとしてよく用いられるアヤメデータを用います。
アヤメデータにはいくつかの変数がありますが、GGally::ggpairs()で一気に視覚化・相関係数を算出してみます。これぞ業務効率化。
library(GGally)
p <- GGally::ggpairs(iris, aes(colour=Species))
p
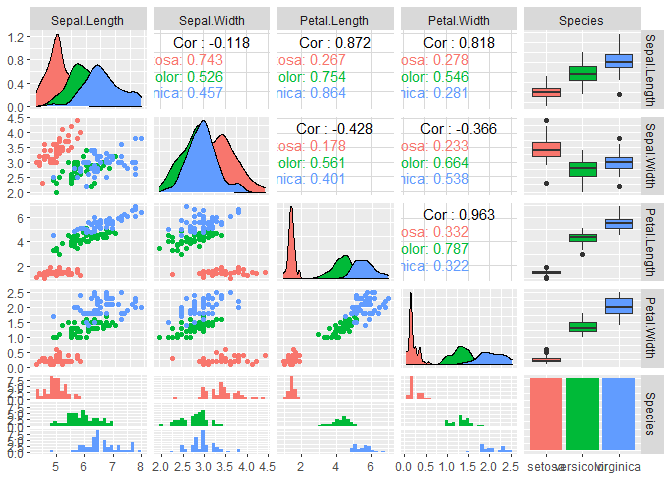
結果を見ると、花びらの長さ(Peral.Length)と幅(Petal.Width)には強い正の相関(0.963)があることが分かります。他にも様々な関係性が確認できますが、以降では花びらの長さを説明するためのモデルを構築していきます。
モデル1
はじめは最も単純なモデルから。まずは花びらの幅のみで花びらの長さを説明する式です。
ここで、
上の式の意味を見てみましょう。
まず、これまで全く説明していませんでしたが、
また
正規分布の確率密度関数
よって、
さらに
ちなみに先に挙げた式を用いて平均0、標準偏差1の正規分布の確率密度関数を視覚化すると、以下のようになります。
library(ggplot2)
x <- seq(-5,5,0.1)
y <- (1/sqrt(2*pi))*exp(-x^2/2)
p <- ggplot(data=data.frame(X=x,Y=y)) + theme_light()+
geom_line(aes(x=X,y=Y)) + xlab(NULL) + ylab(NULL)
p
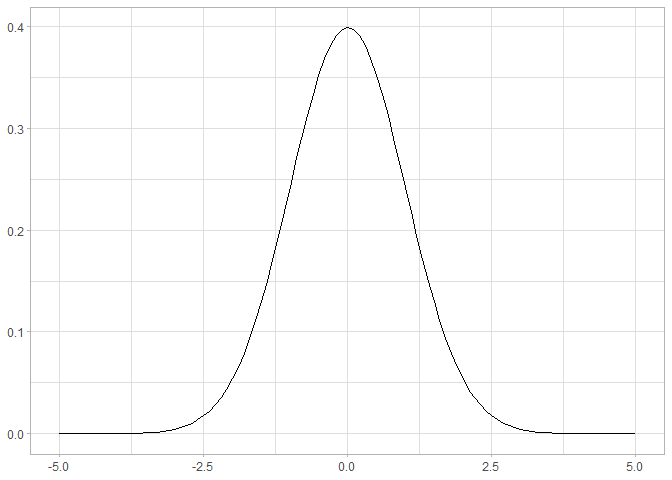
このように、正規分布は左右対称であり、平均と標準偏差自体がパラメータとなる使いやすい分布であるため、様々な統計解析に活用されます。最もよく用いられるのは、今回の例のように連続型変数の誤差の分布を仮定するときではないだろうか?
glm()を用いてこのモデルのパラメータを推測した結果が以下になります。
library(ggeffects)
res <- glm(data=iris, formula = Petal.Length ~ Petal.Width, family=gaussian)
summary(res)
##
## Call:
## glm(formula = Petal.Length ~ Petal.Width, family = gaussian,
## data = iris)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -1.33542 -0.30347 -0.02955 0.25776 1.39453
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.08356 0.07297 14.85 <2e-16 ***
## Petal.Width 2.22994 0.05140 43.39 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## (Dispersion parameter for gaussian family taken to be 0.2286808)
##
## Null deviance: 464.325 on 149 degrees of freedom
## Residual deviance: 33.845 on 148 degrees of freedom
## AIC: 208.35
##
## Number of Fisher Scoring iterations: 2
res_p <- ggeffects::ggpredict(res, terms="Petal.Width")
ggplot() + theme_light(base_size=11) +
geom_point(data=iris, aes(x=Petal.Width, y=Petal.Length), shape=1) +
geom_line(data=res_p, aes(x=x, y=predicted)) +
geom_ribbon(data=res_p, aes(x=x, ymin=conf.low, ymax=conf.high), alpha=0.1)
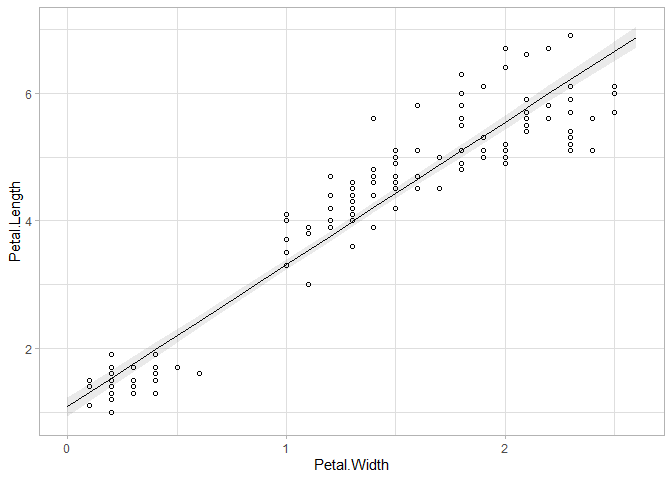
結果がいろいろと書いてありますが、今回はCoefficientsのところから、構築したモデルに対する以下の結果を確認するのみとします(Devianceなどについて復習するのが面倒になった顔)。信頼区間の算出方法も実はよくわかっていません…。ベイズ的なアプローチならこのあたりの理論を事後分布で回避できるのに…
ここまで読んで、エクセルのグラフ機能についている近似曲線(線形近似)と何が違うのか、と思ったかもしれません。実は以上の分析はエクセルでもは全く同じ結果が得られるはずです。違うのは解析手法(最小二乗法か最尤法)くらいかもしれません。しかし単に線形補完をしているのだ、という認識ではなく、上のように統計的なモデルが背景にあるということを理解していると、データに対してより適切な仮定を置くことが可能になります。
はっきり言いますが、ガウス分布を仮定した線形回帰の場合、①目的変数はすべての実数を定義域に持つ連続変数、②目的変数の誤差が正規分布に従うと仮定するため、これらの条件を満たしたデータでないとガウス分布を仮定した線形回帰の結果を信頼することはできない。ということ!
上記の仮定を無視した例としては、離散型変数を目的変数に置いたり、ガウス分布を仮定した線形回帰をあてはめた際の誤差の分布をQQプロットや正規性の検定等で確認しない場合が挙げられます。
モデル2
モデル1は単回帰と呼ばれ、一つの説明変数しか設定しない分析でしたが、説明変数は一つである必要はありません。より多くの情報を持たせた方がモデルの精度が向上することも十分あります。複数の説明変数が存在する場合、重回帰と呼ばれます。
GGally::ggpairs()の結果からアヤメの種類(Species)も花びらの長さに影響しそうだと判断し、説明変数に先ほどの花びらの長さに加えてアヤメの種類(Species)を追加してみます。モデル式は以下になります。
説明変数に
res <- glm(data=iris, formula = Petal.Length ~ Petal.Width + Species, family=gaussian)
summary(res)
##
## Call:
## glm(formula = Petal.Length ~ Petal.Width + Species, family = gaussian,
## data = iris)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -1.02977 -0.22241 -0.01514 0.18180 1.17449
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.21140 0.06524 18.568 < 2e-16 ***
## Petal.Width 1.01871 0.15224 6.691 4.41e-10 ***
## Speciesversicolor 1.69779 0.18095 9.383 < 2e-16 ***
## Speciesvirginica 2.27669 0.28132 8.093 2.08e-13 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## (Dispersion parameter for gaussian family taken to be 0.1426948)
##
## Null deviance: 464.325 on 149 degrees of freedom
## Residual deviance: 20.833 on 146 degrees of freedom
## AIC: 139.57
##
## Number of Fisher Scoring iterations: 2
res_p <- ggeffects::ggpredict(res, terms=c("Petal.Width[all]","Species"), rawdata=T)
library(RColorBrewer)
col=brewer.pal(3,"Set1")
ggplot() + theme_light(base_size=11) +
geom_point(data=iris, aes(x=Petal.Width, y=Petal.Length, colour=Species), shape=1) +
geom_line(data=res_p, aes(x=x, y=predicted, colour=group)) +
geom_ribbon(data=res_p, aes(x=x, ymin=conf.low, ymax=conf.high, fill=group), alpha=0.3) +
scale_fill_manual(values=col) + scale_color_manual(values=col)
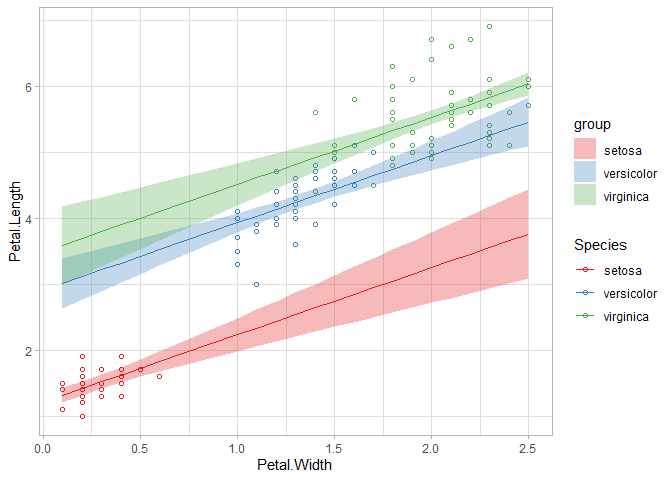
推定結果は以下の通りです。
このように、モデリングではある変数を説明するための変数を追加したり取り除いたりし、結果を確認しながら最終的にどの変数を用いるか検討していきます。
モデル3
モデル1およびモデル2は、目的変数の誤差に正規分布を仮定したもので、一般線形モデルとも呼ばれるモデルでした。しかし目的変数の性質によっては誤差が正規分布でかていできない場合もあります。その場合に有効なのが一般化線形モデル(GLM)です。GLMでは誤差に正規分布だけでなく二項分布やポアソン分布、負の二項分布など様々な分布を一般化して扱います。
今回の例の場合、花びらの長さは負の値をとらないことから厳密には正規分布を仮定できません。そこで非負の値を定義域に持つ連続変数に適用できるガンマ分布を誤差が従う分布に仮定します。
モデル式は以下になります。ガンマ分布はshapeパラメータ
res <- glm(data=iris, formula = Petal.Length ~ Petal.Width + Species, family=Gamma(link="log"))
summary(res)
##
## Call:
## glm(formula = Petal.Length ~ Petal.Width + Species, family = Gamma(link = "log"),
## data = iris)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -0.34682 -0.05608 -0.00637 0.05856 0.28685
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.31996 0.01716 18.643 < 2e-16 ***
## Petal.Width 0.24062 0.04005 6.008 1.43e-08 ***
## Speciesversicolor 0.80728 0.04760 16.960 < 2e-16 ***
## Speciesvirginica 0.90676 0.07400 12.253 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## (Dispersion parameter for Gamma family taken to be 0.009874458)
##
## Null deviance: 44.6546 on 149 degrees of freedom
## Residual deviance: 1.4566 on 146 degrees of freedom
## AIC: 93.256
##
## Number of Fisher Scoring iterations: 4
res_p <- ggeffects::ggpredict(res, terms=c("Petal.Width[all]","Species"), rawdata=T)
library(RColorBrewer)
col=brewer.pal(3,"Set1")
ggplot() + theme_light(base_size=11) +
geom_point(data=iris, aes(x=Petal.Width, y=Petal.Length, colour=Species), shape=1) +
geom_line(data=res_p, aes(x=x, y=predicted, colour=group)) +
geom_ribbon(data=res_p, aes(x=x, ymin=conf.low, ymax=conf.high, fill=group), alpha=0.3) +
scale_fill_manual(values=col) + scale_color_manual(values=col)
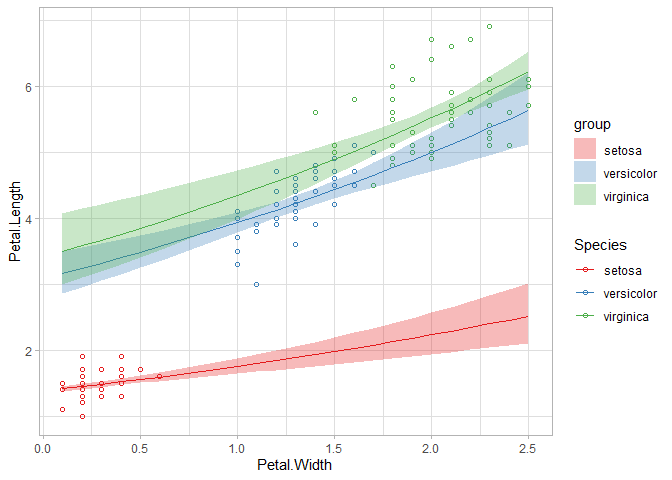
推定結果は以下の通りです。
結果の解釈はお任せしますが、モデルの背景にある仮定をデータの特徴に合わせてより適切なものに修正した形になります。
まとめ
今回はモデリングとは?ということで記事を書いてみました。まとめると以下のような作業がデータの生成過程を組み立てる一連の活動を構成するのかな、と思います。
- 変数の取捨選択
- 確率変数やその誤差が従う分布を仮定
次はモデルの取捨選択を手助けする情報量基準についてまとめてみようかなとおもっています。
